+49 (0) 3727 / 58-1242
Bestes Poster auf dem Molecular Biophysics Meeting der DGfB
Am 17. Juni 2022 fand das Molecular Biophysics Symposium der Deutschen Gesellschaft für Biophysik (DGfB) als Online-Veranstaltung mit rund 80 Teilnehmenden statt. Die Biophotonik aus Mittweida war neben dem Vortrag von Prof. Richard Börner auch durch das Poster "FRET-assisted modeling pipeline for a ribosomal tertiary contact" von Felix Erichson vertreten, der damit den Preis für das beste Poster gewonnen hat. Herzlichen Glückwunsch!
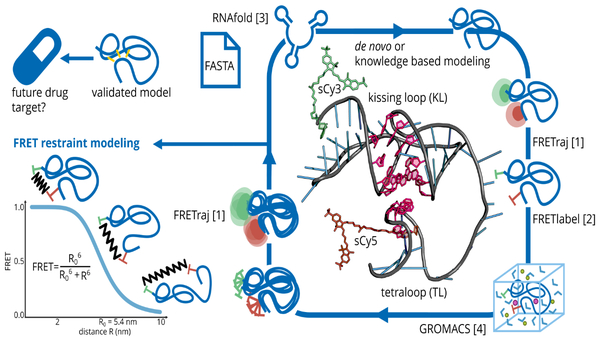
Felix Erichson ist wissenschaflticher Mitarbeiter in der Forschungsgruppe Einzelmolekülspektroskopie von RNA und entwickelt ein Software-Framework für die FRET-gestützte Strukturvorhersage von Ribonukleinsäuren. Dieses Framework besteht aus unabhängigen Software-Komponenten, die vor allem für Anfänger aufgrund der Vielzahl an Parametern und der unterschiedlichen Ansteuerung (von command line bis drag and drop) schwer zu bedienen sind. Dieses Framework soll so weiterentwickelt werden, dass anhand einer Nukleinsäure-Sequenz und experimenteller Parameter, wie dem Förster Resonanz-Energietransfer, kurz FRET, einem Abstandsmesser auf Molekülebene (nm), auf die 3D-Struktur zurückgeschlossen werden kann. Dabei steht zunächst eine kürzlich beschriebene Struktureinheit der ribosomalen RNA der Bäckerhefe im Fokus. Diese Struktur besteht aus drei Bausteinen: zwei RNA-Haarnadelschleifen (engl. hairpins), die eine Kussschleife (engl. kissing loop) bilden und ein GAAA Motif (engl. Tetraloop). Durch die Interaktion dieser Bausteine bildet sich ein Tetraloop-Rezeptor. Diese Bausteine werden in silico als PyMOL-basiertes RNA-Lego verbunden, die Struktur mit Farbstoffen markiert und so das Minimalkonstrukt konstruiert. Das Minimalkonstrukt kann so auch im FRET-Experiment untersucht werden. Die im Computer vorhergesagte durchschnittliche FRET-Effizienz stimmt schon heute sehr gut mit dem FRET-Experiment überein. Dieser hybride Ansatz aus Experiment und Simulation wird als sogenannte integrative RNA-Modellierung nicht nur das Verständnis der RNA-Strukturbildung erweitern, sondern der am LHM entwickelte Ansatz wird dazu beitragen, die Entdeckung neuartiger RNA-Strukturen zu revolutionieren. Das Konzept birgt großes Potential in der medizinischen Diagnostik und der Suche nach neuen Medikamenten.